DNASTAR软件利用PacBio HiFi测序 技术改进基因组组装
DNASTAR公司有时会收到关于使用PacBio HiFi测序进行DNA组装的问题。因此我们决定写一篇关于这个主题的文章。A部分将以问答形式呈现,方便您快速找到您感兴趣的信息。B部分将是一个逐步演示,展示如何在Lasergene软件中设置和分析PacBio HiFi测序的组装结果。
Part A: PacBio HiFi Q&A
在送样进行测序时,为什么可能会选择PacBio HiFi测序,而不是其他长读长或短读长测序技术呢?
PacBio HiFi测序是一种尖端的DNA测序技术,能够提供高准确性的长读长测序。
传统的“短读长”测序方法产生的DNA片段相对较短,而PacBio HiFi测序则能够生成更长的片段,通常可达数万碱基对的长度。这对于解析复杂的基因组区域(如重复序列和结构变异)至关重要。
HiFi测序的读长可以达到高达30kb,并且具有很高的准确性(大于99.9%),与Sanger测序相当。这对于在高同源性和重复性区域进行可靠的变异检测和基因组组装至关重要,因为这些区域是短读长测序技术无法准确比对的。此外,虽然短读长技术可以用于准确检测单核苷酸多态性(SNPs)和小的插入/缺失(indels),但HiFi测序能够准确检测更广泛的变异类型,包括结构变异、相位变异以及甲基化模式。
同样地,尽管短读长测序技术被广泛用于RNA-seq基因表达分析,但要解析全长mRNA转录本却很困难,而HiFi测序则可以用于解析全长mRNA转录本,并精确分类可变剪接事件。
在处理HiFi数据时,常见的陷阱或挑战有哪些,如何避免这些问题?
我没有发现任何特定于HiFi数据的问题,这些问题不也是短读长数据所面临的。这两种数据类型都可能产生大量的数据,需要强大的数据存储和计算资源。
为了最大化HiFi测序读取质量和产量,文库制备和测序的最佳实践是什么?
为了最大化每个SMRT Cell的HiFi产量,PacBio建议将基因组DNA片段化,使其大小分布模式在15 kb到18 kb之间,用于人类全基因组测序。不推荐使用大小分布模式超过20 kb的文库进行HiFi测序。
HiFi技术的未来发展方向和潜在应用是什么?
未来将得到扩展的一个应用领域是临床宏基因组学。目前,临床诊断依赖于培养方法,这些方法可能无法检测到低丰度细菌的存在,或者那些难以在培养基上生长的细菌。相比之下,HiFi宏基因组测序不依赖于细菌在培养基上的生长能力,能够为临床样本提供一种成本效益高且全面的微生物学分析。
HiFi数据与其他长读长技术在准确性、成本和通量方面有何比较?何时会选择一种而不是另一种?

一般来说,HiFi测序的准确性更高(>99.9%),但与Nanopore测序相比,其读长较短(最长25Kb),成本也更高。Nanopore的读长可以超过1Mb,且在高通量潜力方面可能更具成本效益。当需要高准确性时,例如在解析复杂的基因组区域或单倍型相位时,HiFi是最佳选择;而Nanopore可能更适合于大规模项目或从头组装,其中最长的读长可以跨越难以解析的重复区域,且更具成本效益。
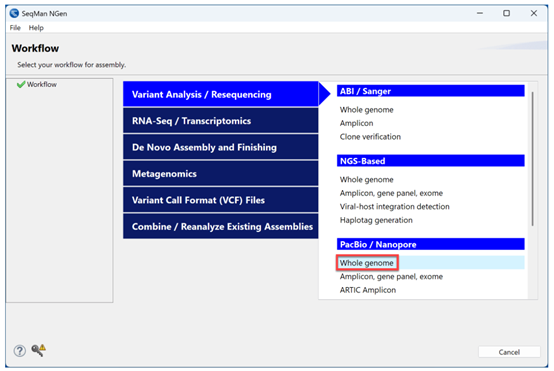
哪些SeqMan NGen工作流程通常使用PacBio HiFi?例如,我可以将其用于转录组分析或宏基因组学吗,还是它仅用于全基因组/外显子组组装?
SeqMan NGen支持PacBio HiFi数据的从头组装和参考引导的组装与比对。在Lasergene 18.0中,支持微生物的从头基因组组装以及参考引导的基因组和外显子组比对。
在SeqMan NGen中使用HiFi数据时,可以对杂合变异进行分相吗?如果可以,该如何操作?在哪里分析结果?
是的,SeqMan NGen提供了一种新的单倍型分相算法,可以检测分相变异。分析工作是在GenVision Pro中完成的,可以在其中可视化分相区域(分相块)以及它们包含的变异。此外,还可以用不同颜色显示单个分相序列读取,以便识别分相块区域内的杂合等位基因。
在SeqMan NGen中组装HiFi数据的计算要求是什么(例如,内存、CPU、GPU、磁盘空间)?是否有基于云的选项?
SeqMan NGen的当前版本(18.0版)使用CPU、空闲磁盘空间和内存(RAM)的组合来比对PacBio HiFi数据,具体需求会根据数据集的大小而变化。对于人类基因组大小的数据,最佳性能需要8核及以上的CPU、32GB内存以及一个专门的4TB硬盘来处理临时文件。SeqMan NGen的下一次更新(18.1版)将能够利用GPU处理,这将大大加快HiFi数据的组装速度,并消除对4TB空闲磁盘空间的需求。也有一些基于云的选项,允许用户在本地设置HiFi组装,然后让数据自动压缩、上传到云端,并在亚马逊云硬件上进行组装(之后会自动下载)。对于希望并行处理多个数据集,或者没有足够强大的本地计算资源来处理大型组装项目的用户来说,这是一个很好的选择。
如何管理和存储HiFi测序产生的大数据集?
你需要很大的存储空间。我的个人电脑是一台强大的i7惠普笔记本电脑,配备了两个硬盘(2TB和4TB)。我还会使用外部4TB硬盘来存储额外的大数据集。
Part B:在Lasergene中的组装设置和下游分析
PacBio HiFi数据(以及其他所有长读长类型)都可以用于从头组装以及变异分析/重测序组装。以下示例展示了如何为果蝇(Drosophila melanogaster)数据集设置并分析一个参考引导的组装。
在SeqMan NGen中设置并运行组装
1)启动SeqMan Ngen并点击 New Assembly。
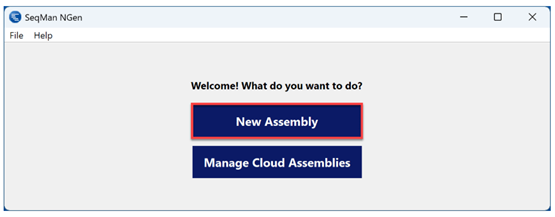
2)在SeqMan NGen Workflow界面下Variant Analysis / Resequencing选项卡中,选择PacBio / Nanopore
Whole genome
3)在Reference Sequence界面中,点击Download Genome Package,从DNASTAR选择一个经过策划的模板包。
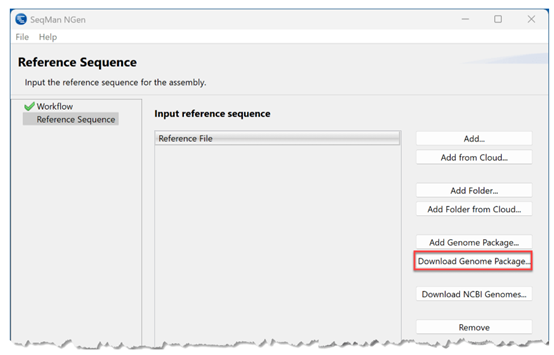
选择Drosophila melanogaster然后点击Select.
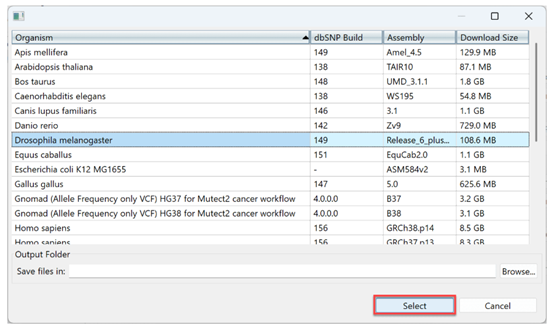
4)在Input Sequences界面中,选择PacBio HiFi作为读取技术。通过点击Add按钮加载PacBio HiFi序列(此处为一个50MB的.fastq文件),或者像图中所示,从文件资源管理器中拖拽它并放到SeqMan NGen向导界面上。
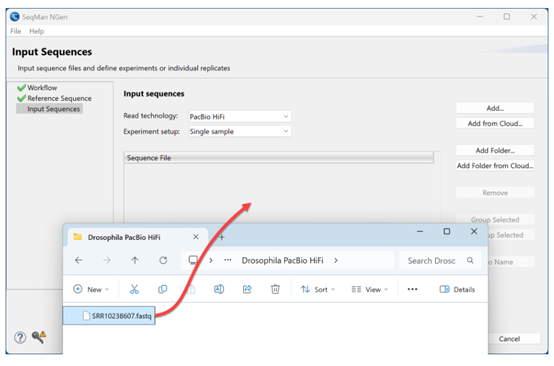
5)连续点击Next两次,到达Analysis Options 界面。默认情况下,Detect SNPs and other small variants已被勾选。由于该生物是二倍体,我们将选择可选步骤Diploid – Phased。这会使SeqMan NGen在组装过程中将变异按等位基因分开,以便我们在分析时可以查看分相变异。由于我们不知道果蝇的性别,因此选择Unknown。
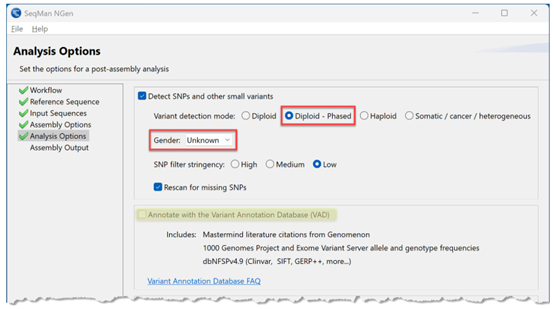
(顺便提一下,如果我们处理的是人类样本,并且希望在组装过程中自动为发现的变异添加增强注释,我们会在第3步中选择人类参考基因组版本37或38。然后,在这一步中,我们会勾选Annotate with the Variant Annotation Database这个选项,如上图中黄色高亮所示。)
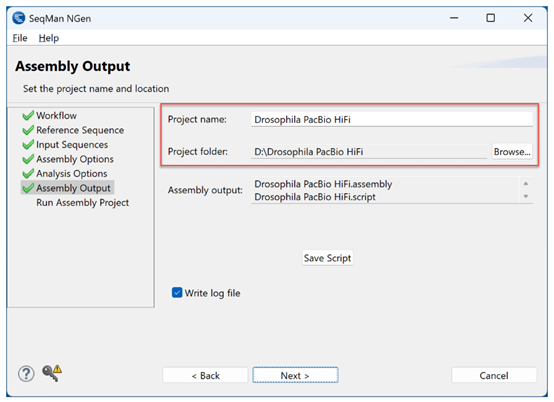
7)单击Next到 Run Assembly Project界面。在这种情况下,SeqMan NGen已经检查了我们计算机的可用内存,并建议在本地运行该程序集。因此,我们点击Run assembly on this computer链接以启动程序集。
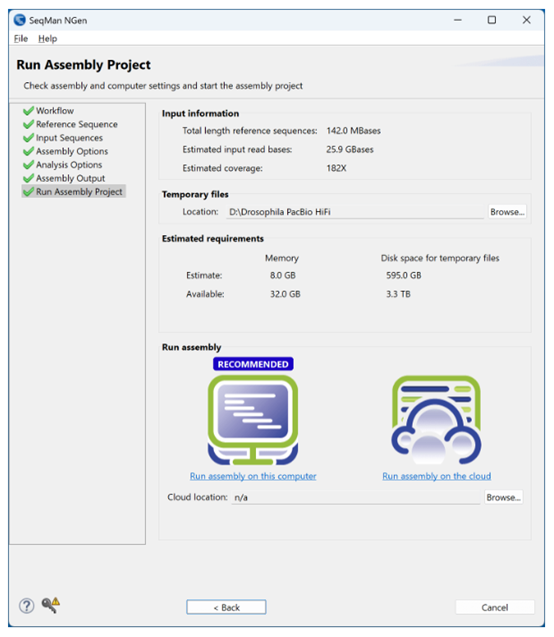
8)一旦装配完成,“XNG done”字眼将会出现在装配日志的底部,并且在右下角会出现一个激活的Finish按钮。
9) 点击Next,来到Assembly Summary界面。
10)若要在GenVision Pro中打开装配文件,请点击Analyze and compare variants按钮。
在GenVision Pro中分析变体
在Genome Pro中,左侧的基因组视图将每条染色体显示为一行交替的蓝色和绿色相位块,而右侧的实验面板则列出了染色体及其长度。
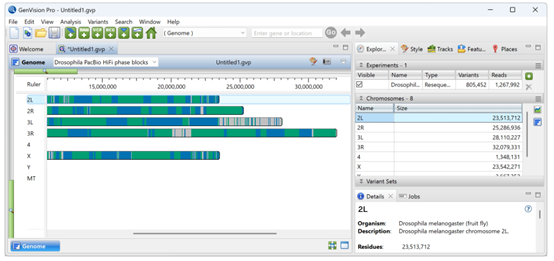
1)要在分析视图中更详细地查看某条染色体,只需在其所在行上双击,无论是在哪个位置。当处于放大状态时,会显示特征和相位块的高级视图。在下面的图像中,您可以清楚地看到绿色相位块的结束位置和蓝色相位块的开始位置。对比色的垂直线表示这些位置上的变异。
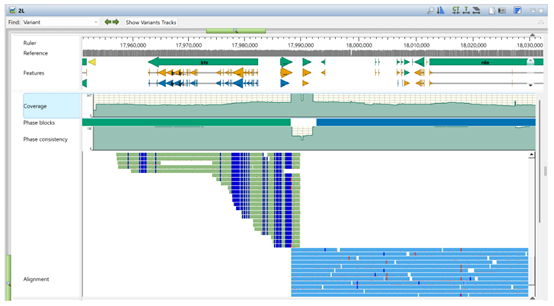
2)按下右上角的CT或T工具以放大并查看单个碱基和变异。在下面的图像中,位置18558809处有一个变异,它位于Pde11基因内。参考序列行在该位置显示为“T”。在比对轨迹中,绿色显示的等位基因也有一个“T”,但蓝色显示的等位基因有一个变异“A”。在那下面,变异轨迹以图形方式显示了相同的内容。请注意,变异轨迹是分叉的,并且在一个等位基因中显示了一个变异(短竖线),而在另一个等位基因中没有显示。

3)要创建一个变异表格,点击实验面板中的 Show Variants Table工具。
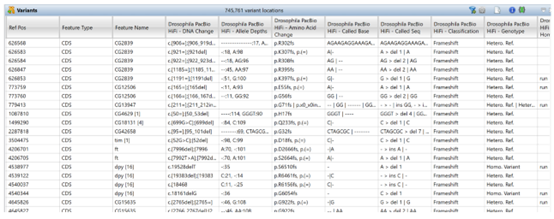
众多可定制的数据列提供了关于每个变异的统计数据和其他有价值的信息。在下面的变异表格图像中,仅展示了这些列的一个子集。
结论
PacBio HiFi的高准确性和长读长能力正在改变基因组学研究。Lasergene的SeqMan NGen 通过直观的设置界面、内置变异检测以及如单倍型分型等功能,简化了此类数据的组装和分析。对于下游分析,GenVision Pro 提供了多种视图和众多定制化选项,包括强大的变异过滤功能。
热点资讯
- 2025-02-06汇聚“银发”力量 ,再绽“时代芳华”!青岛定陶路小学举行老教协2025迎新会
- 2025-08-16鉴赏: 一枚九百多克的老熟俄籽
- 2025-07-07三条道路开工!合肥再添交通“大动脉”_大皖新闻 | 安徽网
- 2024-10-14许家印被捕后,恒大歌舞团长低调嫁人,深扒发现其丈夫身份不简单
- 2025-02-02日本乒乓名将丹羽孝希因涉嫌参与跨境赌博被查
- 2025-07-05DNASTAR软件利用PacBio HiFi测序 技术改进基因组组装
相关资讯
- 汇聚“银发”力量 ,再绽“时代芳华”!青岛定陶路小学举行老教协2025迎新会
- 许家印被捕后,恒大歌舞团长低调嫁人,深扒发现其丈夫身份不简单
- DNASTAR软件利用PacBio HiFi测序 技术改进基因组组装
- “中国白·德化瓷”妇女儿童主题作品展落地中国妇女儿童博物馆
- 日本乒乓名将丹羽孝希因涉嫌参与跨境赌博被查

